Aplicação de método ensemble para classificação de imagens médicas cardíacas
Application of ensemble method for classification of cardiac medical images
Aplicación del método ensemble para la clasificación de imágenes médicas cardíacas
DOI: 10.59681/2175-4411.v17.2025.1207
Guilherme Ormond Sampaio
Bacharel em Ciência da Computação pelo Centro Universitário FEI.
https://orcid.org/0009-0003-5007-6839
Vinícius Dutra
Bacharel em Ciência da Computação pelo Centro Universitário FEI.
https://orcid.org/0009-0006-5359-5433
Leila Cristina Carneiro Bergamasco
Professora no Centro Universitário FEI.
https://orcid.org/0000-0002-8001-8053
Autor Correspondente: Leila Cristina Carneiro Bergamasco
Artigo recebido: 29/07/2024 | Aprovado: 12/08/2025
RESUMO
As doenças cardíacas são responsáveis por cerca de 17 milhões de mortes globalmente, segundo a Organização Mundial da Saúde. Esse cenário tem motivado um aumento na busca por acompanhamento médico preventivo, o que resultou em uma maior demanda por exames de ressonância magnética cardíaca. A identificação de cardiomiopatias nesse volume crescente de exames representa um desafio significativo para as equipes médicas. Com o objetivo de auxiliar nesse processo, este trabalho propõe uma metodologia baseada em aprendizado de máquina supervisionado para o reconhecimento de cardiomiopatias. O método considera diferentes slices do órgão e leva em conta as especificidades do ciclo cardíaco, abordando limitações de abordagens anteriores. Durante os experimentos, foi obtida uma acurácia de 80,00% e uma precisão de 82,26% no melhor cenário de teste, que considera as estruturas do epicárdio e do endocárdio durante a fase diastólica do ciclo cardíaco. Os resultados demonstram o potencial da abordagem proposta no apoio ao diagnóstico médico, especialmente em contextos de alta demanda por exames.
Descritores: aprendizado de máquina; cardiomiopatias; cinerressonância magnética.
ABSTRACT
Heart diseases are responsible for approximately 17 million deaths worldwide, according to the World Health Organization. This scenario has led to an increased demand for preventive medical care, resulting in a higher number of cardiac magnetic resonance imaging (MRI) exams. Identifying cardiomyopathies within this growing volume of exams poses a significant challenge for medical teams. To support this process, this study proposes a supervised machine learning methodology for the recognition of cardiomyopathies. The method takes into account different slices of the heart and the specific characteristics of the cardiac cycle, addressing limitations found in previous approaches. During the experiments, an accuracy of 80.00% and a precision of 82.26% were achieved in the best test case, which considers the structures of the epicardium and endocardium during the diastolic phase of the cardiac cycle. The results highlight the potential of the proposed approach in supporting medical diagnosis, especially in contexts of high exam demand.
Descriptors: machine learning; cardiomyopathies; magnetic resonance imaging; cine.
RESUMEN
Las enfermedades cardíacas son responsables de aproximadamente 17 millones de muertes en todo el mundo, según la Organización Mundial de la Salud. Este panorama ha llevado a un aumento en la búsqueda de atención médica preventiva, lo que ha resultado en una mayor demanda de exámenes de resonancia magnética cardíaca. La identificación de cardiomiopatías dentro de este volumen creciente de estudios representa un desafío significativo para los equipos médicos. Con el objetivo de apoyar este proceso, este trabajo propone una metodología basada en aprendizaje automático supervisado para el reconocimiento de cardiomiopatías. El método considera diferentes cortes del órgano y las especificidades del ciclo cardíaco, abordando las limitaciones de enfoques anteriores. Durante los experimentos, se obtuvo una precisión del 80,00% y una exactitud del 82,26% en el mejor caso de prueba, el cual considera las estructuras del epicardio y el endocardio durante la fase diastólica del ciclo cardíaco. Los resultados demuestran el potencial del enfoque propuesto para apoyar el diagnóstico médico, especialmente en contextos con alta demanda de exámenes.
Descriptores: aprendizaje automático; cardiomiopatías; imagen por resonancia cinemagnética.
INTRODUÇÃO
De acordo com dados divulgados pela Organização Mundial da Saúde, doenças cardíacas são a principal causa de morte em todo o mundo. Essa situação permanece por mais de duas décadas, alcançando a marca de aproximadamente 9 milhões de mortes entre 2000 e 2019(1). Esse cenário, acompanhado do crescimento populacional, ocasionou um aumento na busca por acompanhamento médico e, consequentemente, por exames de diagnóstico.
Um exemplo desse tipo de exame é a ressonância magnética cardíaca (RMC), que registra imagens da anatomia e funcionamento do coração, possibilitando sua utilização na identificação da presença ou ausência de várias doenças cardíacas, entre elas a doença de cardiomiopatia. Tais exames possuem alta resolução e geram dados complexos, características que tornam seu armazenamento e processamento uma tarefa onerosa para os sistemas computacionais. Para lidar com esse desafio, estratégias e técnicas para realizar o processamento desse volume considerável de dados foram desenvolvidas e, atualmente, são ainda objetos de estudo por pesquisadores.
Embora exames de RMC apresentem imagens nítidas e detalhadas sobre a doença de cardiomiopatia, nota-se que em situações no qual a patologia não é explícita, ou seja, não há uma deformação considerável na estrutura cardíaca causada pela doença, a análise desse exame demanda um esforço considerável do profissional responsável, sendo suscetível a falhas humanas que podem interferir na obtenção de um diagnóstico preciso(2). A Figura 1 ilustra exames com casos extremos das diferentes cardiomiopatias, já a Figura 2 apresenta situações nos quais também há a presença de cardiomiopatias, porém as diferenças entre os exames não são tão evidentes.
Nesse contexto, estratégias de classificação utilizando modelos baseados em aprendizado de máquina supervisionado figuram entre as principais abordagens para lidar com este desafio e auxiliar os especialistas durante a elaboração dos diagnósticos. Entre esses modelos, destacam-se o support vector machines (SVM), k-nearest-neighbors (KNN), random forest e redes neurais convolucionais (CNN)(3). No entanto, as informações utilizadas para realizar essa análise, em sua maioria, não consideram as especificidades de cada etapa do ciclo cardíaco(4-5).
Figura 1: Diferentes tipos de cardiomiopatia com patologia explícita: a) cardiomiopatia dilatada e b) cardiomiopatia hipertrófica.
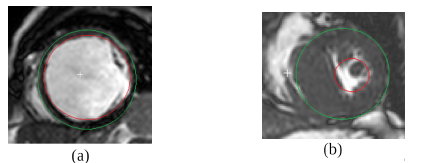
Fonte: Bergamasco, L. 2018
Figura 2: Diferentes tipos de cardiomiopatia com deformações similares: a) cardiomiopatia dilatada e b) cardiomiopatia hipertrófica.

Fonte: Bergamasco, L. 2018
Dessa forma, o presente artigo visa abordar o desafio envolvendo a identificação e classificação de cardiomiopatias em exames de RMC, explorando a influência das especificidades dos diferentes ciclos cardíacos na assertividade dos algoritmos de aprendizado de máquina.
Para realizar a tarefa de classificação foi proposto um modelo que utiliza aprendizado por voto majoritário contendo a princípio, três modelos base em sua composição. Os algoritmos escolhidos para compor o sistema ensemble foram o SVM, Random Forest e KNN, uma vez que tais algoritmos são amplamente adotados em tarefas classificatórias de imagens médicas(6-7-8)
MATERIAIS
Para o desenvolvimento foi utilizada a linguagem de programação Python para a construção desse projeto. Também foram utilizadas as bibliotecas scikit-learn, que implementa diversos algoritmos de aprendizado de máquina, e a biblioteca pandas, que auxilia na manipulação de bases de dados dentro da linguagem escolhida para a implementação. Para o processamento do modelo foi utilizado um computador com sistema operacional Windows 10, processador Intel i5-8400, 16GB de memória RAM e uma graphics processing unit (GPU) NVIDIA GeForce RTX 2060 com 6GB GDDR3 de memória.
Em relação a base de dados, foi utilizada uma base anonimizada de pacientes com e sem a doença de cardiomiopatia criada e fornecida pelo Instituto do Coração, pertencente ao Hospital das Clínicas da Faculdade de Medicina da Universidade de São Paulo (HCFMUSP – InCor), seguindo as devidas precauções para garantir a ética e anonimidade dos dados, sendo submetido ao CEP (Comitê de Ética e Pesquisa) com o processo de número 2.800.046. Os dados fornecidos são rotulados em pacientes com cardiomiopatia hipertrófica, dilatada ou sem anomalia. Essa base é composta de 112 exames de RMC, sendo composta por 42 exames sem anomalias, 35 com cardiomiopatia hipertrófica e 35 com cardiomiopatia dilatada. Essas imagens foram previamente segmentadas por profissionais do InCor.
A Figura 3 apresenta exemplos de imagens obtidas após a segmentação, a linha verde indica a estrutura do epicárdio e a vermelha, a estrutura do endocárdio, sendo que a área formada entre as duas linhas é denominada parede ventricular. Após segmentação do endocárdio e epicárdio é gerado um arquivo com os valores para todos os pontos cartesianos que compõem as curvas utilizadas na segmentação. Esse processo foi realizado para todo o ciclo cardíaco, desde o momento de máxima contração (sístole) até máxima dilatação (diástole).
Figura 3: Imagens do ventrículo esquerdo com a segmentação da parede ventricular
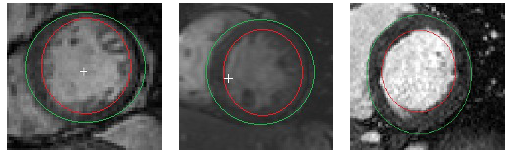
Fonte: Bergamasco, L. 2018
MÉTODOS
A Figura 4 apresenta um diagrama esquemático com a metodologia utilizada nesta pesquisa. A partir das imagens de RMC segmentadas foram calculadas as áreas do endocárdio, epicárdio e, utilizando essas duas medidas, foi possível obter a área da parede ventricular em cada exame analisado. Os descritores são calculados considerando cada etapa do ciclo cardíaco, e a multidimensionalidade dos slices e frames.
Como segunda etapa foram aplicados os classificadores de aprendizado supervisionado SVM, KNN e Random Forest. O classificador SVM tem por objetivo determinar o hiperplano que melhor diferencia os conjuntos de dados utilizando funções de kernel. Já o algoritmo KNN atribui uma categoria para um novo dado a partir da avaliação de seus k vizinhos utilizando uma medida de similaridade, como distância Euclidiana. Dessa forma, o dado será rotulado de acordo com a classificação mais comum entre seus k vizinhos avaliados. Por fim, o classificador Random Forest é um algoritmo de aprendizado supervisionado usado para tarefas de classificação que combina várias árvores de decisão para melhorar a precisão e a robustez do modelo. Durante o treinamento, o algoritmo cria diversas árvores de decisão a partir de diferentes subconjuntos aleatórios dos dados de treinamento. A previsão final do modelo é feita através de uma votação por maioria, onde cada árvore de decisão individual faz uma previsão e a classe escolhida pela maioria das árvores é selecionada como a classificação final do classificador. Essa abordagem aumenta a precisão do modelo e reduz o risco de overfitting, tornando o Random Forest mais robusto e confiável em comparação com uma única árvore de decisão(9).
Em seguida, todas as saídas desses classificadores são organizadas em um sistema ensemble, que determina a classificação final através de voto majoritário(10-11).
Figura 4: Diagrama esquemático com a metodologia aplicada.
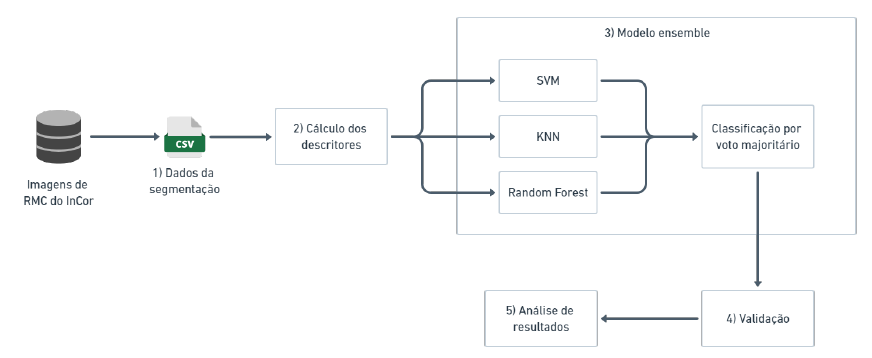
Fonte: Elaborado pelos próprios autores.
Para o classificador SVM foi utilizado o radial basis function kernel (kernel RBF) para definição dos vetores de suporte e do plano de separação, já no classificador KNN foi utilizada a distância de Manhattan como medida de similaridade para estabelecer a classe atribuída ao caso de análise e para o classificador Random Forest foram construídas árvores de decisão para classificar cada possível condição a ser identificada (inexistente, cardiomiopatia hipertrófica ou cardiomiopatia dilatada) e para adicionar diversidade na construção das árvores, é aplicado o método de bootstrap aggregating (bagging) na construção dos conjuntos de dados para treinamento. Finalmente, para realizar o treinamento dos classificadores citados acima é utilizado o método k-fold cross validation, com valor de k igual a 5.
A discussão será então apresentada visando responder as seguintes questões de pesquisa:
QP1: O tipo de estrutura cardíaca pode influenciar nos resultados de classificação?
QP2: Qual ciclo cardíaco apresenta melhores resultados classificatórios?
QP3: Dado que os exames de RMC podem possuir diferentes quantidades de frames segmentados, existe algum intervalo de imagens que apresenta melhores resultados classificatórios?
QP4: O método ensemble pode auxiliar na classificação de cardiomiopatias?
Testes e Validação
Em relação aos testes, foram planejados diferentes casos de teste envolvendo as estruturas segmentadas (epicárdio, endocárdio e parede ventricular) e os ciclos cardíacos (ciclo completo, sístole e diástole). Os casos de teste podem ser validados observados na tabela 1.
Tabela 1: Casos de teste. Tabela completa disponível online.
-
Identificador
Estruturas
Ciclo
Slices
Frames
Ciclos alinhados
CT01
Endocárdio
Completo
Todos
1-30
Não
CT02
Endocárdio
Parcial
Todos
1-16
Não
...
CT50
Endo + PV
Completo
Todos
Picos e média
N/A
CT51
Epi + PV
Completo
Todos
1-30
Não
CT52
Epi + PV
Parcial
Todos
1-16
Não
...
CT69
Todas
Completo
Todos
Picos
Sim
CT70
Todas
Completo
Todos
Picos e média
Sim
Fonte: Elaborado pelos próprios autores.
Dado a discrepância na quantidade de frames presente nos exames constituintes, natureza do equipamento de condução do exame, diversas estratégias de preenchimento foram testadas para garantir a maior eficácia do modelo de aprendizado de máquina. A tabela 2 apresenta o resumo das estratégias.
Tabela 2: Estruturação dos cenários de teste
-
Cenários
Ciclos
Preenchimento das áreas
Limitados
Completos
Zero
Médias no final
Médias no início
Cenário 1
x
x
Cenário 2
x
x
Cenário 3
x
x
Cenário 4
x
x
Cenário 5
x
x
Cenário 6
x
x
Fonte: Elaborado pelos próprios autores.
“Ciclos Limitados” significa restringir o número de frames utilizados para o ciclo em questão. Isto é, caso no cenário o ciclo diastólico seja limitado a dez frames, mesmo que ele possua dezesseis em sua totalidade, serão considerados apenas os primeiros dez frames. O oposto, denominado “Ciclos Completos”, são definidas a quantidade máxima de frames para o ciclo conforme o maior valor encontrado nos exames avaliados.
A definição das áreas pode ser realizada de três formas: com o valor zero, representando um valor nulo; com o valor médio final da área calculada entre dois frames, utilizando a média entre o último valor e o penúltimo; ou com o valor médio inicial calculado entre os dois frames iniciais.
Para validação foram utilizadas as seguintes métricas: sensitividade, especificidade, precisão, acurácia, F1-score e AUROC. Essas métricas são utilizadas devido ao seu alto emprego em trabalhos relacionados ao tema(12,13). Adicionalmente foram feitas análises descritivas para identificar estruturas e ciclos cardíacos com melhor desempenho nos testes.
RESULTADOS E DISCUSSÃO
Neste capitulo são apresentados e discutidos os resultados obtidos durante o processo experimental.
Os resultados estão ordenados de forma decrescente de acordo com a acurácia obtida após execução do modelo ensemble e estão apresentados nas tabelas 3, 4, 5, 6, 7 e 8.
Tabela 3: Resultados obtidos com modelo o Ensemble no Cenário 1.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT48
79,05%
92,31%
81,68%
79,05%
79,21%
0,895
CT39
78,10%
91,10%
79,53%
78,10%
78,04%
0,878
CT69
77,14%
90,88%
80,39%
77,14%
76,88%
0,882
Fonte: Elaborado pelos próprios autores.
Tabela 4: Resultados obtidos com modelo o Ensemble no Cenário 2.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT48
78,10%
90,99%
80,73%
78,10%
78,25%
0,888
CT36
78,10%
89,89%
80,42%
78,10%
78,02%
0,880
CT32
77,14%
89,78%
79,15%
77,14%
77,01%
0,873
Fonte: Elaborado pelos próprios autores.
Tabela 5: Resultados obtidos com modelo o Ensemble no Cenário 3.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT36
79,05%
89,89%
81,22%
79,05%
78,93%
0,876
CT38
78,10%
90,88%
81,19%
78,10%
77,70%
0,864
CT62
78,10%
91,10%
80,26%
78,10%
78,03%
0,876
Fonte: Elaborado pelos próprios autores.
Tabela 6: Resultados obtidos com modelo o Ensemble no Cenário 4.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT38
79,05%
91,10%
81,74%
79,05%
78,88%
0,872
CT36
78,10%
89,67%
80,68%
78,10%
78,06%
0,878
CT48
78,10%
91,10%
80,33%
78,10%
77,80%
0,878
Fonte: Elaborado pelos próprios autores.
Tabela 7: Resultados obtidos com modelo o Ensemble no Cenário 5.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT38
80,00%
91,21%
82,26%
80,00%
79,94%
0,867
CT36
78,10%
89,89%
80,42%
78,10%
78,02%
0,875
CT32
77,14%
89,78%
79,15%
77,14%
77,01%
0,876
Fonte: Elaborado pelos próprios autores.
Tabela 8: Resultados obtidos com modelo o Ensemble no Cenário 6.
-
Identificador
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
CT39
78,10%
91,21%
80,62%
78,10%
77,95%
0,869
CT36
78,10%
89,89%
80,42%
78,10%
78,02%
0,882
CT38
78,10%
90,99%
79,76%
78,10%
77,83%
0,866
Fonte: Elaborado pelos próprios autores.
A tabela 9 demonstra as estruturas dos casos de teste mencionados nos resultados.
Tabela 9: Variáveis de controle utilizadas nos casos de teste apresentados nos Cenários.
-
Identificador
Estruturas
Ciclo
Slices
Frames
Ciclos alinhados
CT32
Endo + Epi
Parcial
Todos
1-16
Não
CT36
Endo + Epi
Parcial
Todos
1-16
Inı́cio na diástole
CT38
Endo + Epi
Diástole
Todos
N/A
Sim
CT39
Endo + Epi
Completo
Todos
Picos
N/A
CT48
Endo + PV
Diástole
Todos
N/A
Sim
CT62
Todas
Parcial
Todos
1-16
Não
CT69
Todas
Completo
Todos
Picos
N/A
Fonte: Elaborado pelos próprios autores.
Uma característica que se destacou durante a realização dos experimentos foi a combinação das estruturas. Todos os casos de teste que figuraram entre os melhores considerando a acurácia, se utilizaram de uma combinação de estruturas,
nenhum caso que analisou uma estrutura isoladamente figurou entre os cinco melhores em seus respectivos cenários.
Observa-se também uma predominância do ciclo diastólico entre os casos de teste que apresentaram as melhores acurácias.
O caso de teste CT38 nas condições estabelecidas no Cenário 5 (preenchimento de áreas com médias no início e ciclos reduzidos) apresentou a melhor acurácia durante a realização dos experimentos. Nesse caso de teste utilizou-se as estruturas endocárdio e epicárdio, e restringiu-se a análise ao ciclo diastólico.
O mesmo caso de teste se mostrou presente entre os melhores resultados em outros cenários, conforme observado na Tabela 10. Nestes cenários ele apresentou métricas similares ao resultado disposto no Cenário 5, exceto nos Cenários 1 e 2, em que apresentou uma acurácia abaixo de 78,10%.
Tabela 10: Comparativo entre as métricas calculadas no modelo Ensemble sobre o CT38 nos diferentes cenários.
-
Cenários
Métricas
Sensitividade
Especificidade
Precisão
Acurácia
F1-score
AUROC
Cenário 1
74,29%
88,46%
76,38%
74,29%
74,12%
0,874
Cenário 2
73,33%
87,80%
76,53%
73,33%
73,49%
0,872
Cenário 3
78,10%
90,88%
81,19%
78,10%
77,70%
0,864
Cenário 4
79,05%
91,10%
81,74%
79,05%
78,88%
0,872
Cenário 5
80,00%
91,21%
82,26%
80,00%
79,94%
0,867
Cenário 6
78,10%
90,99%
79,76%
78,10%
77,83%
0,866
Fonte: Elaborado pelos próprios autores.
Uma característica que se destacou durante a realização dos experimentos foi a combinação das estruturas. Todos os casos de teste que figuraram entre os melhores de acordo com a acurácia se utilizaram de uma combinação de estruturas. Uma representação visual sobre a distribuição das estruturas entre os melhores casos de cada cenário pode ser vista na Figura 5.
Figura 5: Presença das estruturas nos melhores casos de teste em cada cenário.
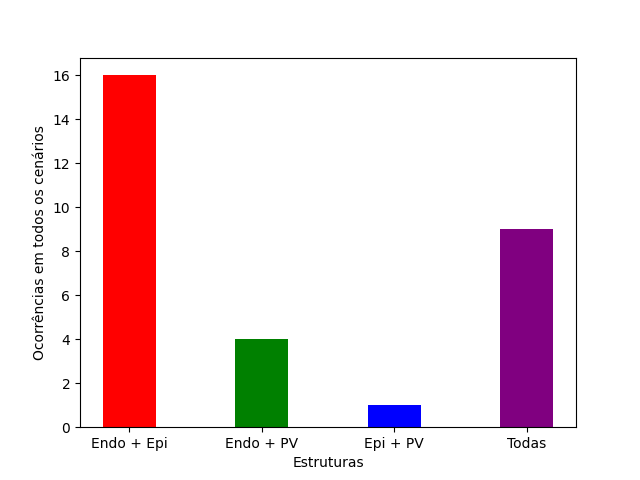
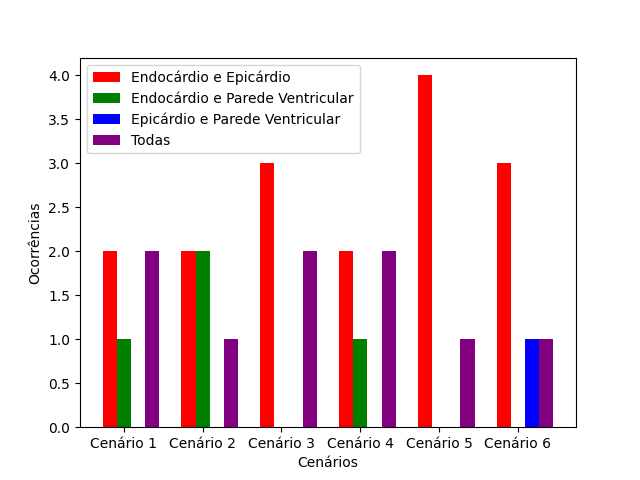
Fonte: Elaborado pelos próprios autores.
Observa-se também uma predominância do ciclo diastólico entre os casos de teste que apresentaram as melhores acurácias, conforme observado na Figura 6. Agregou-se em cada ciclo (diastólico e sistólico) também os casos em que, embora o teste não se restrinja apenas àquele ciclo, seu início possui alinhamento com o ciclo em questão.
Figura 6: Presença dos ciclos nos melhores casos de teste em cada cenário.
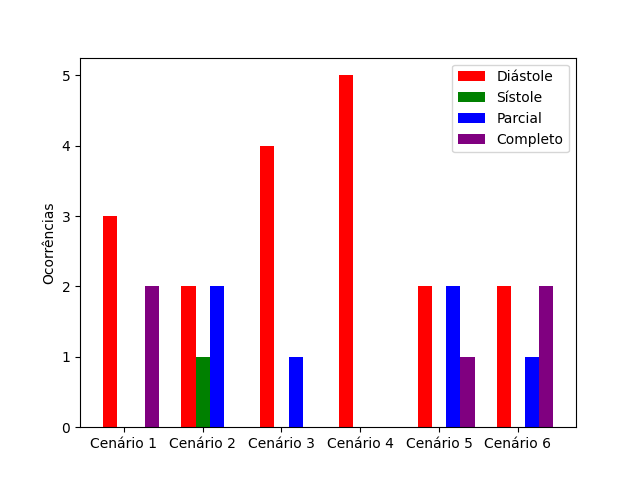
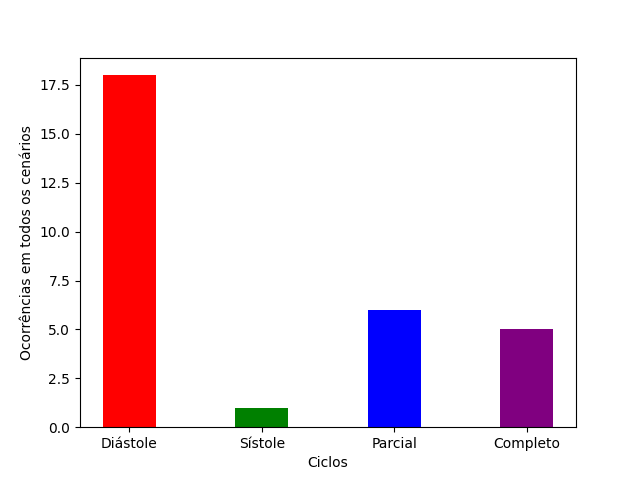
Fonte: Elaborado pelos próprios autores.
Ao se observar o desempenho do modelo Ensemble em cada classificação de forma binarizada (abordagem one vs rest), é possível identificar uma acurácia e precisão maiores para a identificação da cardiomiopatia dilatada, alcançando cerca de 88% em acurácia e precisão. As outras classificações apresentam desempenhos semelhantes entre si, alcançando 83% de precisão e 74% de acurácia.
Embora não foram encontrados na literatura corrente trabalhos que utilizem aprendizado de máquina supervisionado para realizar classificações em condições similares na mesma base, é possível comparar com trabalhos que realizam atividades similares no mesmo conjunto de dados. Sendo assim, comparado ao trabalho de Bergamasco(4) que utilizou a mesma base de dados, porém com outros descritores e em uma tarefa de recuperação e não de classificação, a abordagem proposta na presente pesquisa obteve uma média de precisão 22% superior.
Embora não seja possível comparar diretamente os resultados com o de trabalhos que utilizam outros datasets e exames, é possível obter uma noção de como essa abordagem se situa em comparação às demais. Os seguintes trabalhos utilizam bases de dados de ecocardiogramas, mas focam também na classificação de cardiomiopatias dilatadas e hipertróficas: o trabalho de Nasimov(14) utiliza a base de dados EchoNet-Dynamic, e obteve 96,4% de acurácia; o trabalho de Balaji(15) obteve uma acurácia ligeiramente menor, 92,04%, e possuem um dataset próprio.
CONCLUSÃO
Neste trabalho foi proposto um modelo para identificar e classificar cardiomiopatias no ventrículo esquerdo, utilizando descritores que avaliam a área das estruturas do ventrículo, obtidas através de segmentações em exames de RMC.
Utilizando um modelo que combina as classificações do SVM, KNN e Random Forest através de voto majoritário, registrou-se uma acurácia de 80% e precisão de 82,26% ao utilizar-se das estruturas epicárdio e endocárdio, limitar a avaliação ao ciclo diastólico e restringir o ciclo a treze frames, obtendo uma precisão superior à encontrada na literatura, conforme disposto em Bergamasco(4).
Durante a realização do projeto, notou-se uma restrição relacionada ao número de amostras disponíveis de cada classificação possível. Esse fator restringiu as opções, tanto de modelos a serem empregados, quanto de testes a serem elaborados.
Outro fator limitante foi o arquivo de entrada utilizado no software que realiza as segmentações. Essa necessidade impossibilitou a utilização de outras bases (por exemplo, ACDC), visto que não seria possível obter um arquivo com o exame segmentado de forma similar ao utilizado atualmente. Como apresentado nos resultados, a abordagem utilizada exibe uma performance inferior às que utilizam exames de ecocardiograma, esses resultados denunciam e reforçam a escassez de bases de dados de cine-MRI, assim como falta de trabalhos que exploram esses exames para o diagnóstico de cardiomiopatias.
Em relação às questões de pesquisa indicadas na introdução, obteve-se as seguintes conclusões a partir dos experimentos realizados:
QP1: O tipo de estrutura cardíaca pode influenciar nos resultados de classificação? Sim, notou-se que a combinação dos dados referentes a área do epicárdio e endocárdio se sobressaíram às demais possíveis combinações;
QP2: Qual ciclo cardíaco apresenta melhores resultados classificatórios? Foi identificado que ciclo diastólico apresenta os melhores resultados considerando a acurácia. Esse ciclo é o mesmo utilizado por especialistas médicos durante o diagnóstico visual, o que corrobora os resultados obtidos;
QP3: Dado que os exames de RMC podem possuir diferentes quantidades de frames segmentados, existe algum intervalo de imagens que apresenta melhores resultados classificatórios? Não foi possível identificar um conjunto de frames específicos que contribuem significativamente na classificação. As estruturas e o ciclo cardíaco foram mais determinantes;
QP4: O método ensemble pode auxiliar na classificação de cardiomiopatias? Sim, comparado com trabalhos que utilizaram a mesma base, até o momento, foi o método que melhor classificou os casos;
Dito isso, a fim de avançar as pesquisas sobre o tema abordado, sugere-se a realização de estudos para expandir a quantidade de amostras de cada classificação no conjunto de dados utilizados.
Visando a expansão das opções possíveis de amostras para a realização de testes, sugere-se a construção de um processo independente para realizar a segmentação de exames de RMC e que gere arquivos de segmentação semelhantes aos utilizados neste projeto.
Considerando o desempenho dos modelos componentes, recomenda-se também realizar experimentos adotando outros modelos de classificação no modelo ensemble, ou substituir os modelos adotados neste projeto, a fim de estudar o comportamento de outras abordagens utilizando a estratégia de composição de classificadores para essa tarefa.
REFERÊNCIAS
1. World Health Organization (WOS). Cardiovascular diseases. [Internet]. 2021. [citado 2025 ago 13] Disponível em: https://www.who.int/news-room/fact-sheets/detail/cardiovascular-diseases-(cvds)
2. Hanna TN, Zygmont ME, Peterson R, Theriot D, Shekhani H, Johnson JO, et al. The Effects of Fatigue From Overnight Shifts on Radiology Search Patterns and Diagnostic Performance. J Am Coll Radiol. 2018 Dec;15(12):1709-1716. doi: 10.1016/j.jacr.2017.12.019. Epub 2018 Feb 1. PMID: 29366599; PMCID: PMC6054573.
3. Simran V, Gupta A. Effective prediction of heart disease using data mining and machine learning: A review. In International Conference on Artificial Intelligence and Smart Systems (ICAIS); 2021. p. 249-253.
4. Bergamasco L. Recuperação de objetos médicos 3D utilizando harmônicos esféricos e redes de fluxo. Tese de Doutorado. São Paulo: Universidade de São Paulo (USP); 2018.
5. Ghosh P, Azam S, Jonkman M, Karim A, Shamrat F, Ignatious E, et al. Efficient Prediction of Cardiovascular Disease Using Machine Learning Algorithms With Relief and LASSO Feature Selection Techniques. 2021. IEEE Access, 9, 19304-19326.
6. Salvatore C, Cerasa A, Castiglioni I, Gallivanone F, Augimeri A, Lopez M, et al. Machine learning on brain MRI data for differential diagnosis of Parkinson's disease and Progressive Supranuclear Palsy. Journal of Neuroscience Methods. 2014; 222:230-237.
7. Moreno A, Rodriguez J, Martínez F. Regional Multiscale Motion Representation for Cardiac Disease Prediction. XXII Symposium on Image, Signal Processing and Artificial Vision (STSIVA) 2019 (pp. 1-5).
8. Zhuang J, Cai J, Wang R, Zhang J, Zheng WS. Deep kNN for Medical Image Classification. Medical Image Computing and Computer Assisted Intervention – MICCAI 2020. 2020;127–36.
9. Norvig, Russel P, Artificial S. Intelligence. A modern approach. Vol. 90. Prentice Hall, 2020.
10. Raza K. Improving the prediction accuracy of heart disease with ensemble learning and majority voting rule [Internet]. ScienceDirect. Academic Press; 2019. p. 179–96. [citado 2025 ago 13] Disponível em: https://www.sciencedirect.com/science/article/abs/pii/B9780128153703000086
11. Miao K, Miao J, Miao G. Diagnosing Coronary Heart Disease using Ensemble Machine Learning. International Journal of Advanced Computer Science and Applications. 2016;7(10).
12. Fries JA, Varma P, Chin-Hung Chen V, Xiao K, Tejeda H, Saha P, et al. Weakly supervised classification of aortic valve malformations using unlabeled cardiac MRI sequences. Nature Communications. 2019 Jul 15;10(1).
13. Qayyum A, Anwar SM, Awais M, Majid M. Medical image retrieval using deep convolutional neural network. Neurocomputing. 2017 Nov;266:8–20.
14. Nasimov R, Nasimova N, Botirjon K; ABDULLAYEV, Munis. Deep Learning Algorithm for Classifying Dilated Cardiomyopathy and Hypertrophic Cardiomyopathy in Transport Workers. Lecture Notes In Computer Science, [S.L.], p. 218-230, 2023. Springer Nature Switzerland. [citado 2025 ago 13] Disponível em: http://dx.doi.org/10.1007/978-3-031-30258-9_19.
15. Balaji GN, Subashini TS, Chidambaram N. Detection and diagnosis of dilated cardiomyopathy and hypertrophic cardiomyopathy using image processing techniques. Engineering Science And Technology, An International Journal, [S.L.], v. 19, n. 4, p. 1871-1880, dez. 2016. Elsevier BV. [citado 2025 ago 13] Disponível em: http://dx.doi.org/10.1016/j.jestch.2016.10.001.





